全长转录组,是基于三代测序平台,无需打断拼接,直接获得包含5’,3’UTR,polyA尾的完整转录本,从而准确分析有参考基因组物种可变剪接及融合基因等结构信息,克服无参考基因组物种转录本拼接较短、信息不完整的难题。同时还可以借助二代测序数据,进行转录本特异性表达分析,获得更加全面的注释信息。
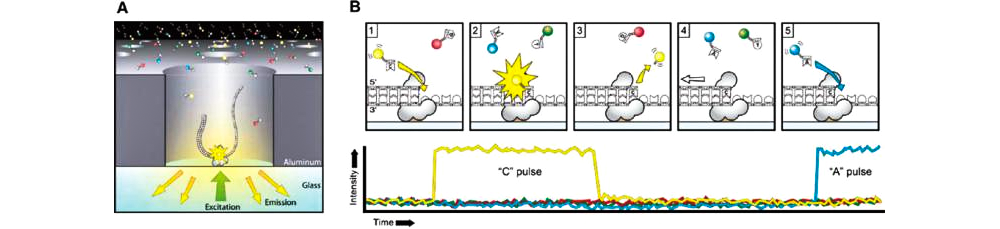
Sequel 测序原理
全长有参转录组:对于有参考基因组的物种,利用三代全长转录组测序获得的信息是生物体内直接存在的,比基于参考基因组预测到的转录组信息更准确,同时也可准确鉴定基因的可变剪接、融合基因、基因家族和非编码RNA等信息。同时基于全长转录组数据,可以优化基因结构,进而辅助基因组组装和注释。
全长无参转录组:由于基因组测序成本高,缺乏基因组参考信息在很大程度上限制了对物种的深入研究。通过三代全长转录组测序来构建物种Unigene库,无需进行序列组装,就可以获得该物种转录组水平的全长转录本信息,更准确的进行CDS和SSR分析,为后续研究提供良好的遗传信息基础。

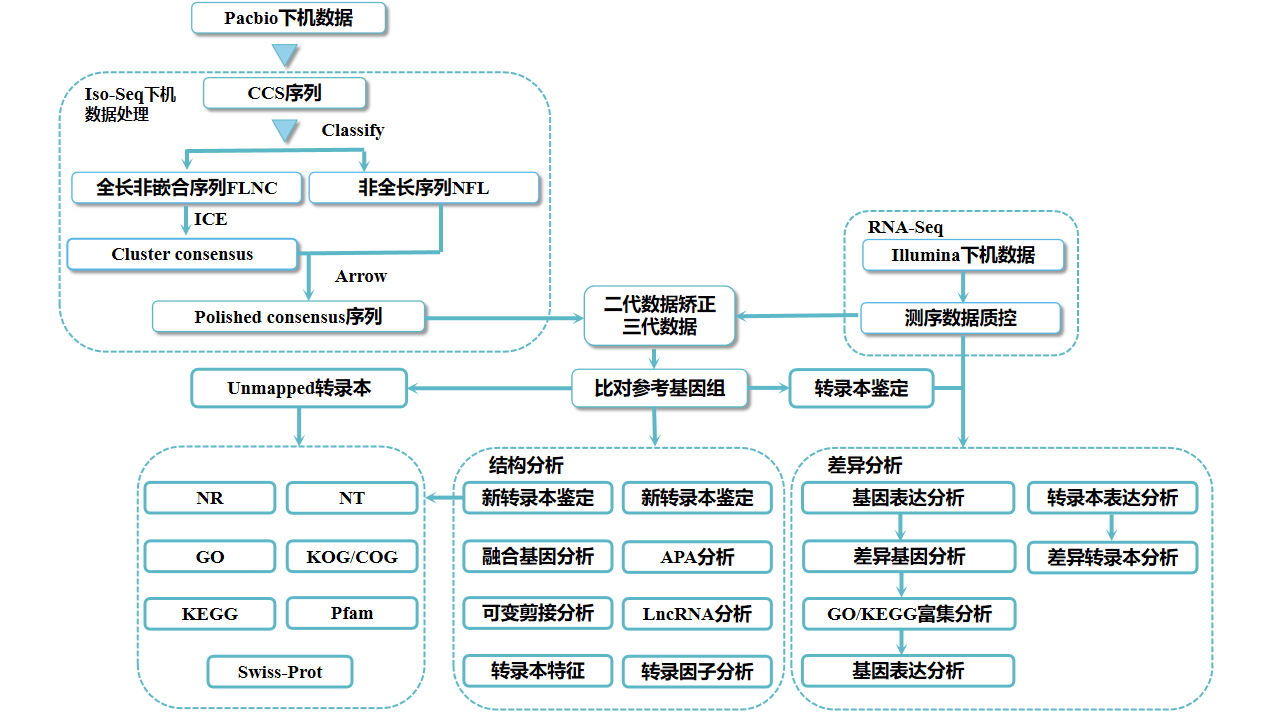
三代全长有参转录组
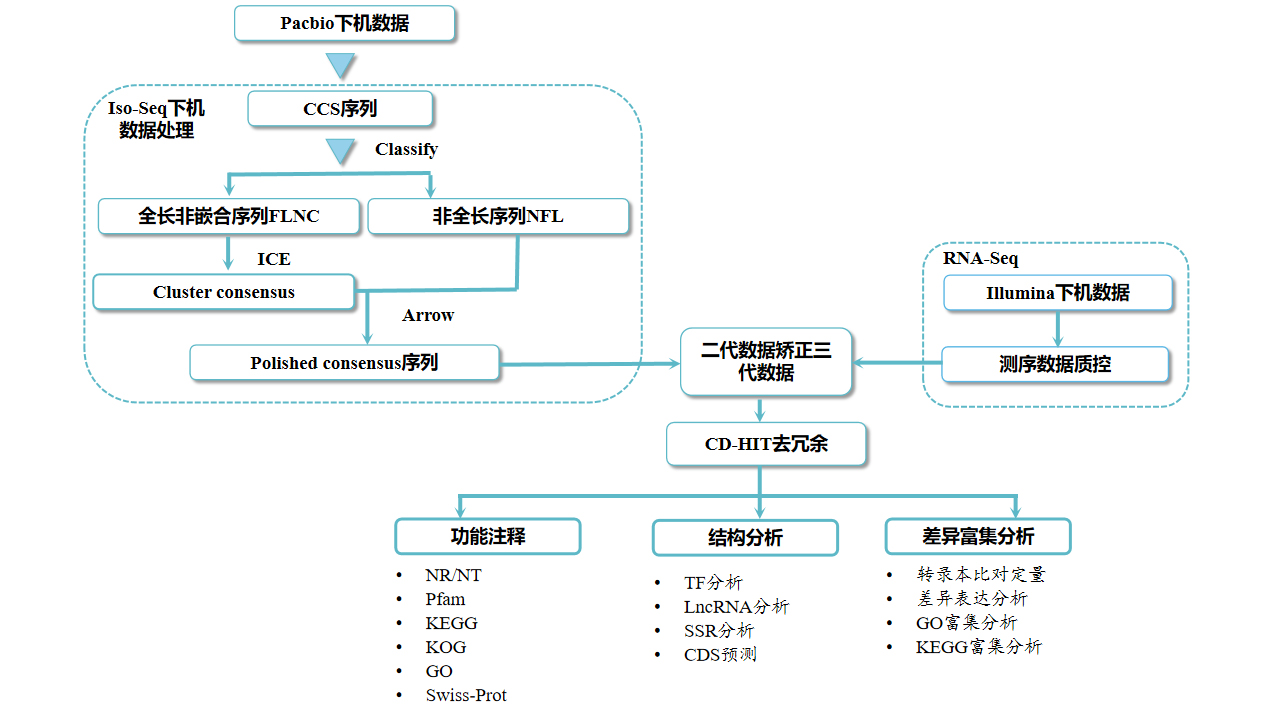
三代全长无参转录组
样品类型 | 总RNA、组织样; |
样品浓度 | 总RNA浓度≥ 50 ng/ul; |
样品体积 | > 10 ul; |
样品总量 | 总RNA用量> 10 ug ; |
样品纯度 | OD260/280≥1.8,OD260/230≥1.0,260nm处有正常峰值; |
RNA完整性 | 总RNA的RIN值≥8.0;图谱基线无上抬;5S峰正常。 |
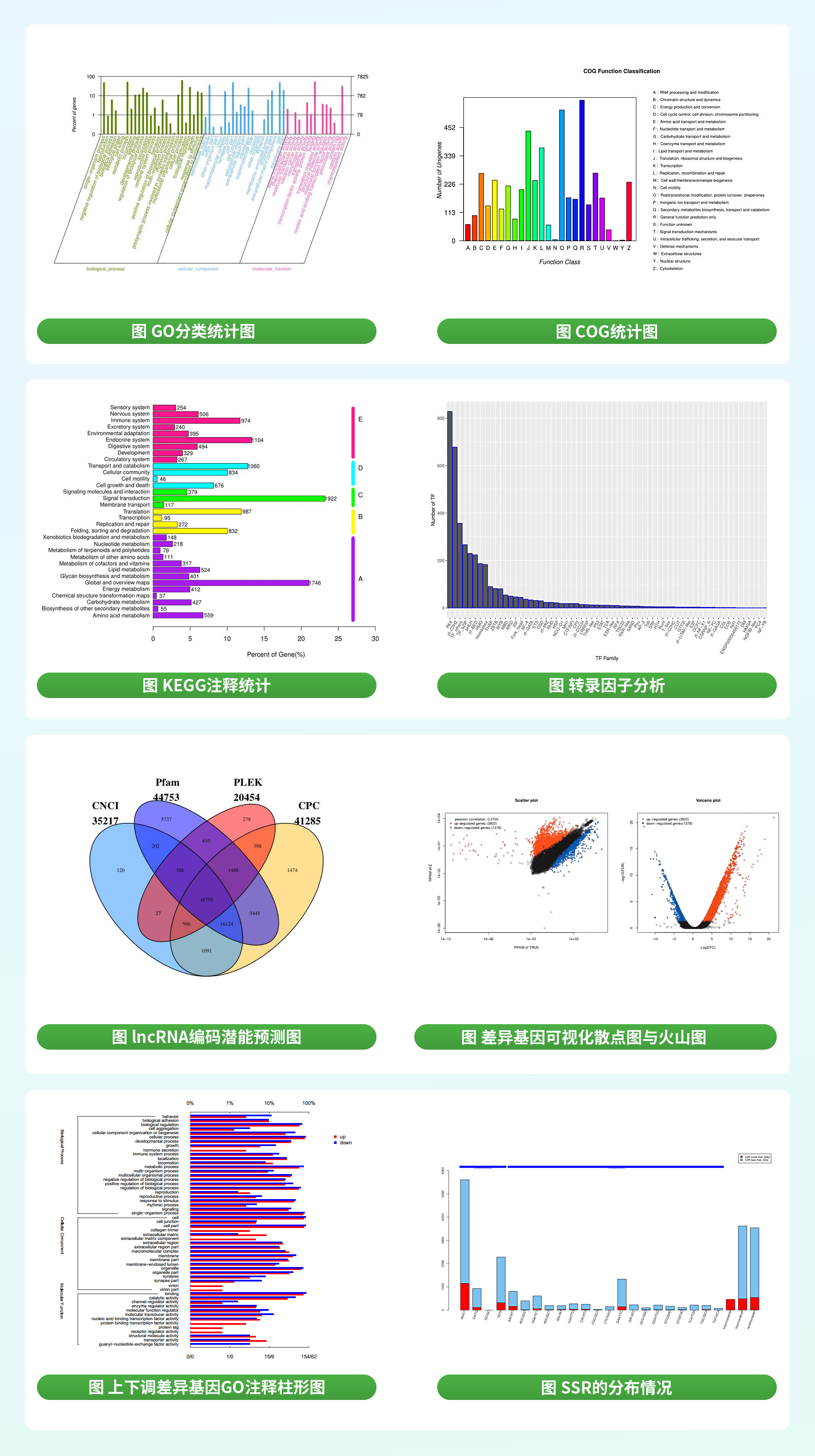
| 发表时间 | 文章标题 | 杂志名 | 影响因子 | 合作单位 |
| 2023 | Full-length transcriptome analysis provides new insights into the diversity of immune-related genes in the threatened freshwater shellfish Solenaia oleivora
| Fish and Shellfish Immunology | 4.7 | 中国水产科学 研究院淡水渔业 研究中心 |
| 2022 | Combined Illumina and Pacbio sequencing technology on transcriptome analysis reveals several key regulations during the early development of American shad (Alosa sapidissima) | Aquaculture Reports | 3.216 | 南京农业大学 |
| 2022 | Full-Length Transcriptome Characterization and Comparative Analysis of Chosenia arbutifolia | Forests | 2.633 | 江苏省林业 科学院 |






